Hochauflösende laserinduzierte Fluoreszenzspektroskopie
Einführung
Molekülstrukturen im elektronischen Grundzustand können mit einer Vielzahl etablierter Techniken wie Mikrowellenspektroskopie, Röntgenbeugung, Neutronenbeugung oder NMR-Spektroskopie bestimmt werden. Geometrien von elektronisch angeregten Zuständen sind viel schwieriger zu erhalten. In unserer Gruppe wird die rotationsaufgelöste Fluoreszenzspektroskopie verwendet, um die Rotationskonstanten des untersuchten Moleküls zu extrahieren. Aus den Spektren können weitere Informationen gewonnen werden, wie Zentrifugalverzerrungskonstanten, Orientierung des Übergangsdipolmoments oder Lebensdauer des angeregten Zustands. Weitere komplementäre Techniken, die auch Strukturinformationen zu elektronisch angeregten Zuständen liefern, sind die resonante Ionisationsvariante der rotationsaufgelösten vibronischen Spektroskopie, die in der Gruppe von Neusser in München entwickelt wurde, und die Rotationskohärenzspektroskopie, die von Felker eingeführt und in der Gruppe von Brutschy und Riehn in Frankfurt. Für die Rotationsauflösung der Spektren großer Moleküle ist eine Auflösung von 1 Teil in 108 über den gesamten UV-Bereich erforderlich.
Experimentell
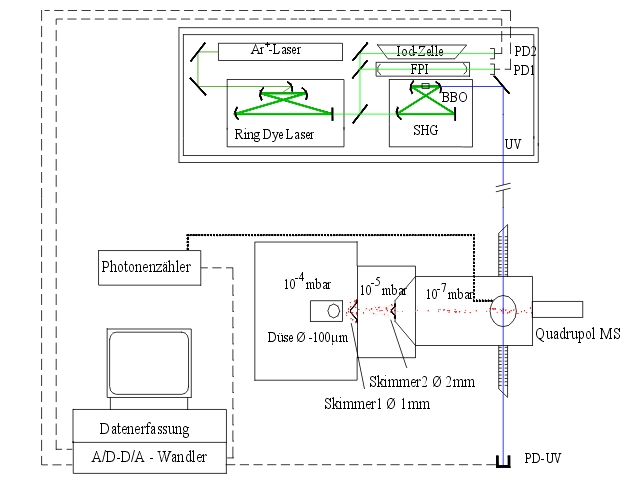
Der Versuchsaufbau für die rotationsaufgelöste laserinduzierte Fluoreszenz (LIF) besteht aus einem Einfrequenz-Ringfarbstofflaser, der entweder mit 6 W der 514 nm Linie eines Ar+ Lasers oder mit 7 W der 515 nm Linie eines diodengepumpten cw Yb:YAG Lasers (ELS MonoDisk-515). Das Licht wird in einen externen gefalteten Ringhohlraum zur Erzeugung der zweiten Harmonischen (SHG) eingekoppelt. Der Molekularstrahl wird gebildet, indem eine meist erhitzte Probe, die typischerweise in 200 - 1000 mbar Argon geimpft wird, durch ein 100 - 300 µm großes Loch in das Vakuum expandiert wird. Die Molekularstrahlmaschine besteht aus drei differentiell gepumpten Vakuumkammern, die durch zwei Skimmer (1 mm bzw. 3 mm) linear verbunden sind, um die Dopplerbreite zu reduzieren. Der Molekularstrahl wird in der dritten Kammer rechtwinklig mit dem Laserstrahl 360 mm hinter der Düse gekreuzt. Die resultierende Fluoreszenz wird senkrecht zu der durch Laser und Molekularstrahl definierten Ebene von einer Abbildungsoptik bestehend aus einem konkaven Spiegel und zwei plankonvexen Linsen gesammelt. Die resultierende Dopplerbreite in diesem Aufbau beträgt 15 MHz (FWHM). In einigen Experimenten betrug die Dopplerbreite 25 MHz, wegen einer etwas anderen Anordnung des optischen Systems. Die integrierte Molekularfluoreszenz wird von einer Photomultiplierröhre detektiert, deren Ausgabe von einem Photonenzähler diskriminiert und digitalisiert und zur Datenaufzeichnung und -verarbeitung an einen PC übertragen wird. Die relative Frequenz wird mit einem kalibrierten quasi-konischen Fabry-Perot-Interferometer mit einem freien Spektralbereich (FSR) von ca. 150 MHz bestimmt. Die absolute Häufigkeit wird durch Aufzeichnen des Jodabsorptionsspektrums und Vergleich der Übergänge mit den tabellarischen Linien bestimmt.
Beispiel: Phenol-Wasser
Der Phenol-Wasser-Cluster dient als Modellsystem für intermolekulare Wasserstoffbrücken zwischen einem polaren Aromaten und einem Lösungsmittelmolekül.
Die Analyse des Spektrums zeigt eine Aufspaltung in zwei Teilbänder aufgrund der inneren Bewegung des Wasseranteils. Die Barriere für diese Torsion konnte mit 180 cm-1 im elektronischen Grund und 130 cm-1 im elektronisch angeregten Zustand bestimmt werden. Aus einer Analyse der Rotationskonstanten konnte festgestellt werden, dass die Struktur translinear ist, mit einer leichten Verkürzung der Wasserstoffbrücke bei elektronischer Anregung.
G. Berden, W. Leo Meerts, M. Schmitt und K. Kleinermanns Hochauflösende UV-Spektroskopie von Phenol und dem wasserstoffgebundenen Phenol-Wasser-Cluster. J. Chem. Phys. 104, 972 (1995).
Beispiel: Phenol-Methanol
Der Wasserstoffbrücken-Phenol-Methanol-Cluster ist ein interessantes Modellsystem, um das Gleichgewicht zwischen Wasserstoffbrückenbindungen der Hydroxygruppen von Phenol bzw. Methanol und den Van-der-Waals-Wechselwirkungen zwischen der Methylgruppe und dem aromatischen Ring zu untersuchen, die beide die Struktur vom Cluster haben. Da drei Rotationskonstanten nicht ausreichen, um die intermolekulare Struktur vollständig zu charakterisieren, haben wir die Rovibronenspektren mehrerer Isotopomere gemessen. Unten sind die rotationsaufgelösten LIF-Spektren von drei verschiedenen Isotopomeren, unterschiedlich deuteriert in der Phenol- und der Methanoleinheit, dargestellt, die mit der automatischen Anpassungstechnik des genetischen Algorithmus analysiert wurden. Weitere Details finden sich in: Determination of the structures and barriers to hindered internal rotation of the phenol-methanol cluster in the S0 and S1 state; Chem. Phys. 254 (2000) 349 - 361.
Beispiel: Das Phenoldimer
Das Phenoldimer ist ein ideales Modellsystem, um das sehr empfindliche Gleichgewicht zwischen Wasserstoffbrückenbindung und Dispersionswechselwirkung zu untersuchen. Die rotationsaufgelösten UV-Spektren der elektronischen Ursprünge von fünf Isotopomeren des Phenoldimers wurden gemessen. Die komplexen Spektren werden mit einer auf genetischen Algorithmen basierenden Anpassungsstrategie analysiert. Aus den Trägheitsparametern wurden die Parameter der intermolekularen Geometrie für beide elektronischen Zustände bestimmt und mit den Ergebnissen von Ab-initio-Rechnungen verglichen. Im elektronischen Grundzustand findet man eine größere Wasserstoffbrückenlänge als in den Ab-initio-Rechnungen und einen kleineren Neigungswinkel der aromatischen Ringe, was eine ausgeprägtere Dispersionswechselwirkung zeigt. Im elektronisch angeregten Zustand nimmt die Wasserstoffbrückenlänge ab, wie bei anderen wasserstoffbrückengebundenen Phenolclustern festgestellt wurde, und die beiden aromatischen Ringe sind weniger zueinander geneigt. Weitere Details sind zu finden in: Schmitt, M., Böhm, M., Ratzer, C., Krügler, D., Kleinermanns, K., Kalkman, I., Berden, G. and Meerts, WL: Electronic excitation in the phenol dimer: The intermolecular structure in the S0 and S1 state determined by rotationally resolved electronic spectroscopy. ChemPhysChem accepted (2006)
Beispiel: Das Benzonitril-Dimer
Das rotationsaufgelöste UV-Spektrum des elektronischen Ursprungs des Benzonitril-Dimers wurde mit einer auf genetischen Algorithmen basierenden Anpassungsstrategie gemessen und analysiert. Für den elektronischen Grundzustand wird eine C2h-symmetrische Struktur gefunden, in der die permanenten Dipolmomente der Benzonitrilmonomere antiparallel ausgerichtet sind. Es konnte gezeigt werden, dass die Orientierung des Übergangsdipolmoments parallel zur Orientierung in der Monomereinheit ist. Der Abstand zwischen den beiden Monomereinheiten nimmt bei elektronischer Anregung leicht ab und die Symmetrie des Benzonitril-Dimers ändert sich von C2h im elektronischen Grundzustand zu Cs im elektronisch angeregten Zustand. Dieser Symmetriebruch wird wahrscheinlich durch die lokale Anregung von nur einem Benzonitril . verursacht aufgrund der schwachen elektronischen Kopplung zwischen den Clustereinheiten. Weitere Details finden sich in: Schmitt, M., Böhm, M., Ratzer, C., Siegert, S., van Beek, M. und Meerts, WL: Electronic excitation in the benzonitrile dimer: The intermolecular structure in the S0 and S1 state determined by rotationally resolved electronic spectroscopy. J. Mol. Struc. accepted (2006).
Beispiel: Azaindol
7-Azaindol (7AI) wurde in den letzten Jahrzehnten zahlreichen experimentellen und theoretischen Studien unterzogen, hauptsächlich weil das 7-Azaindol-Dimer als Modellsystem für tautomere Prozesse in DNA-Basenpaaren dient. Darüber hinaus ist 7-Azaindol der Chromophor des Aminosäureanalogs 7-Azatryptophan, das verwendet wird, um Tryptophan in Proteinen zu ersetzen. Die Abbildung unten zeigt das hochauflösende LIF-Spektrum des elektronischen Ursprungs von 7AI bei 34630,74 cm-1. Das Spektrum wurde automatisch unter Verwendung eines auf einem genetischen Algorithmus (GA) basierenden Fits zugewiesen, der in [1] - [3] beschrieben ist.
[1] Meerts, W. L., Schmitt, M. und Groenenboom, G.: New applications of the Genetic Algorithm for the interpretation of High Resolution Spectra. Can. J. Chem. 82 (2004), 804.
[2] Meerts, W. L. und Schmitt, M.: A new automated assign and analyzing method for high resolution rotational resolved spectra using Genetic Algorithms. Phys. Scripta 73 (2005), C47.
[3] Schmitt, M., Ratzer, C., Kleinermanns, K. und Meerts, WL: Determination of the structure of 7-azaindole in the electronic ground and excited state using high resolution ultra-violet spectroscopy and an automated assignment based on a genetic algorithm. Mol. Phys. 102 (2004), 1605.
Beispiel: Tryptamin
Tryptamin selbst, aber noch mehr seine Analoga Serotonin (5-Hydroxytryptamin) und Melatonin (5-Methoxy-N-acetyl-tryptamin) sind als Neurotransmitter bekannt. Ihre Konformationsstabilitäten und Präferenzen sind von großer Bedeutung für quantitative Struktur-Aktivitäts-Beziehungen bei Neurotransmitter-Rezeptor-Interaktionen. Da alle biologischen Prozesse in wässriger Umgebung ablaufen, ist die Wechselwirkung mit einer definierten Anzahl von Lösungsmittelmolekülen von großem Interesse. Es stellt sich die Frage, wie viele Wassermoleküle notwendig sind, um die große Vielfalt der energetisch zugänglichen Konformationen an die biologisch aktive(n) zu binden. Das unten gezeigte Spektrum ist eines der beeindruckendsten Beispiele für die Leistungsfähigkeit der auf genetischen Algorithmen basierenden automatisierten Zuordnung. Acht überlappende Isotopomere des Tryptamin-B-Konformers wurden gleichzeitig mit der automatisierten Technik des genetischen Algorithmus angepasst. Weitere Details finden sich in: Schmitt, M., Böhm, M., Ratzer, C., Vu, C., Kalkman, I. und Meerts, W. L.: Structural selection by microsolvation: conformational locking of tryptamine. J. Am. Chem. Soc. 127 (2005), 10356.